Navigation auf uzh.ch
Navigation auf uzh.ch
IMMense ist eine Analyse Pipeline für Whole Genome Sequencing (WGS) Daten. Die Pipeline umfasst das Trimmen der Reads, Assemblierung, Annotation, Resistenzgen- und Virulenzfaktor-Bestimmung, Spezies Annotation sowie die Ausführung Spezies-spezifischer Module, kombiniert mit Qualitätskontrollen vom jedem der Schritte. (Bild 1)
Durch die Implementierung in NextFlow und die Verwendung von Singularity Images ist eine hohe Reproduzierbarkeit und Portierbarkeit gewährleistet.
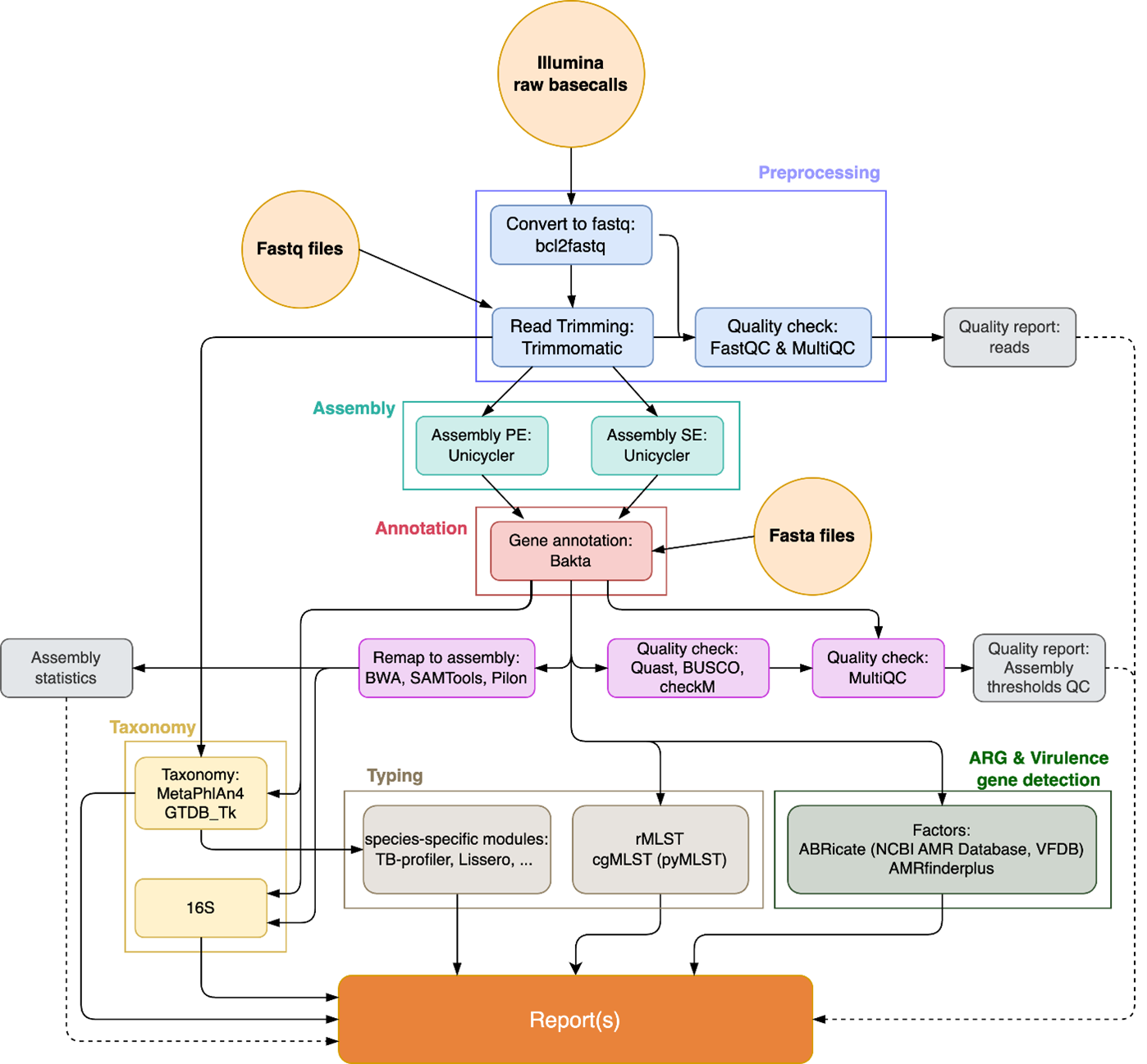
Bild 1: Überblick der IMMense Pipeline. Anhand der Annotation eines Genoms mit MetaPhlan können Spezies-spezifische Module ausgeführt werden.
Für Zugang zum Gitlab Repository können Sie uns per E-Mail kontaktieren: ngs@imm.uzh.ch